函数 dbscan_dynamic_fl() 是一种 UDF(用户定义的函数),它使用 DBSCAN 算法对数据集进行聚类化。 此函数类似于dbscan_fl(),只不过特征由单个数值数组列提供,而不是由多个标量列提供。
先决条件
- 必须在群集上启用 Python 插件。 这是函数中使用的内联 Python 所必需的。
语法
T | invoke dbscan_fl(features_col, cluster_col, epsilon, min_samples, metric, metric_params)
详细了解语法约定。
参数
| 客户 | 类型 | 必需 | 说明 |
|---|---|---|---|
| features_col | string |
✔️ | 包含要用于聚类分析的特征的数值数组的列的名称。 |
| cluster_col | string |
✔️ | 存储每个记录的输出群集 ID 的列的名称。 |
| epsilon | real |
✔️ | 被视为邻居的两个样本之间的最大距离。 |
| min_samples | int |
邻域内一个点被视为核心点的样本数量。 | |
| metric | string |
计算点之间的距离时要使用的指标。 | |
| metric_params | dynamic |
指标函数的额外关键字自变量。 |
函数定义
可以通过将函数的代码嵌入为查询定义的函数,或将其创建为数据库中的存储函数来定义函数,如下所示:
使用以下 let 语句定义函数。 不需要任何权限。
let dbscan_dynamic_fl=(tbl:(*), features_col:string, cluster_col:string, epsilon:double, min_samples:int=10, metric:string='minkowski', metric_params:dynamic=dynamic({'p': 2}))
{
let kwargs = bag_pack('features_col', features_col, 'cluster_col', cluster_col, 'epsilon', epsilon, 'min_samples', min_samples,
'metric', metric, 'metric_params', metric_params);
let code = ```if 1:
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
features_col = kargs["features_col"]
cluster_col = kargs["cluster_col"]
epsilon = kargs["epsilon"]
min_samples = kargs["min_samples"]
metric = kargs["metric"]
metric_params = kargs["metric_params"]
df1 = df[features_col].apply(np.array)
mat = np.vstack(df1.values)
# Scale the dataframe
scaler = StandardScaler()
mat = scaler.fit_transform(mat)
# see https://docs.scipy.org/doc/scipy/reference/spatial.distance.html for the various distance metrics
dbscan = DBSCAN(eps=epsilon, min_samples=min_samples, metric=metric, metric_params=metric_params) # 'minkowski', 'chebyshev'
labels = dbscan.fit_predict(mat)
result = df
result[cluster_col] = labels
```;
tbl
| evaluate python(typeof(*),code, kwargs)
};
// Write your query to use the function here.
示例
以下示例使用 invoke 运算符运行函数。
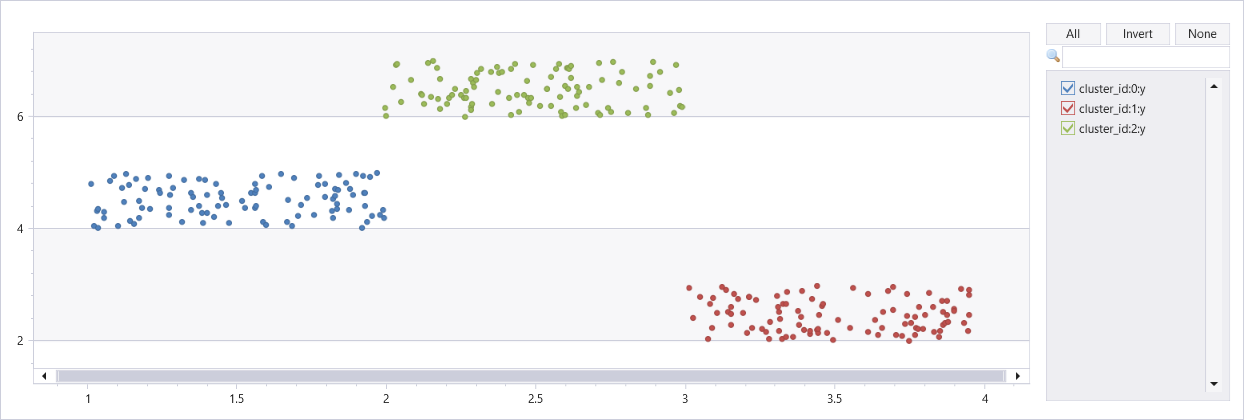
有三个群集的人工数据集群集
若要使用查询定义的函数,请在嵌入的函数定义后调用它。
let dbscan_dynamic_fl=(tbl:(*), features_col:string, cluster_col:string, epsilon:double, min_samples:int=10, metric:string='minkowski', metric_params:dynamic=dynamic({'p': 2}))
{
let kwargs = bag_pack('features_col', features_col, 'cluster_col', cluster_col, 'epsilon', epsilon, 'min_samples', min_samples,
'metric', metric, 'metric_params', metric_params);
let code = ```if 1:
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
features_col = kargs["features_col"]
cluster_col = kargs["cluster_col"]
epsilon = kargs["epsilon"]
min_samples = kargs["min_samples"]
metric = kargs["metric"]
metric_params = kargs["metric_params"]
df1 = df[features_col].apply(np.array)
mat = np.vstack(df1.values)
# Scale the dataframe
scaler = StandardScaler()
mat = scaler.fit_transform(mat)
# see https://docs.scipy.org/doc/scipy/reference/spatial.distance.html for the various distance metrics
dbscan = DBSCAN(eps=epsilon, min_samples=min_samples, metric=metric, metric_params=metric_params) # 'minkowski', 'chebyshev'
labels = dbscan.fit_predict(mat)
result = df
result[cluster_col] = labels
```;
tbl
| evaluate python(typeof(*),code, kwargs)
};
union
(range x from 1 to 100 step 1 | extend x=rand()+3, y=rand()+2),
(range x from 101 to 200 step 1 | extend x=rand()+1, y=rand()+4),
(range x from 201 to 300 step 1 | extend x=rand()+2, y=rand()+6)
| project Features=pack_array(x, y), cluster_id=int(null)
| invoke dbscan_dynamic_fl("Features", "cluster_id", epsilon=0.6, min_samples=4, metric_params=dynamic({'p':2}))
| extend x=toreal(Features[0]), y=toreal(Features[1])
| render scatterchart with(series=cluster_id)